ВУЗ: Не указан
Категория: Не указан
Дисциплина: Не указана
Добавлен: 09.01.2024
Просмотров: 29
Скачиваний: 1
ВНИМАНИЕ! Если данный файл нарушает Ваши авторские права, то обязательно сообщите нам.
Цель контрольной работы: ознакомление с проблемой кластерного анализа при интеллектуальной обработке данных в информационных системах; изучение алгоритмов кластеризации; приобретение навыков в программной реализации изученных алгоритмов на языке Python и в компьютерном проведении кластерного анализа.
Алгоритм CURE
Описание алгоритма кластерного анализа.
В алгоритме CURE выбирается постоянная c точек кластера с хорошим распределением и эти точки стягиваются к центру тяжести кластера на некоторое значение. Точки после стягивания используются как представители кластера. Кластеры с ближайшей парой представителей объединяются на каждом шаге алгоритма иерархической кластеризации CURE.
Шаг 1. Если все данные использовать сразу как входные для CURE, то эффективность алгоритма будет низкая, а время выполнения большим. Поэтому на первом шаге мы случайным образом выбираем часть точек, которые помещаются в память, затем группируем наиболее похожие с помощью иерархического метода в заранее заданное число кластеров. Дальше работаем с кластерами.
Шаг 2. Для каждого кластера выбираем C точек-представителей, максимально удаленных друг от друга. Чисто С остается постоянным.
Шаг 3. Объединяем кластеры с наиболее похожими наборами точек-представителей. Если не достигнуто нужное число кластеров, то перейти на шаг 2. Чтобы при объединении кластеров не выбирать каждый раз c точек-представителей из всех точек, мы выбираем их только из 2С точек объединенных кластеров.
Выбранные точки сдвигаются на следующем шаге на alpha к центроиду кластера. Алгоритм становится основанным на методе поиска центроида при alpha = 1, и основанным на всех точках кластера при alpha = 0.
Первый шаг алгоритма.
1) Присваивание точкам индексов и создание кластеров для каждой из точек
2) Определение ближайших кластеров
Для всех кластеров
Берем найденную на предыдущем запуске цикла дистанцию (или очень большое число при первом запуске)
Находим новую минимальную дистанцию
Присваиваем найденный ближайший кластер тому, для которого вызвана функция
3) Пока число кластеров больше заданного при запуске алгоритма
3.1) Найти индекс кластера, который можно объединить с соседом
Для всех кластеров
Берем найденную на предыдущем запуске цикла дистанцию (или очень большое число при первом запуске)
Находим новую минимальную дистанцию
3.2) Создаем новый кластер из двух соседей
3.3) Пересчет связей в списке кластеров
Для всех кластеров
Дистанция до нового кластера
Если найденная дистанция меньше самой близкой
Объявляем кластер ближайшим соседом нового
Если ближайший кластер текущего был одним из объединённых
Если этот кластер ближе, чем новая минимальная дистанция
Присваиваем ему новый ближайший
Если нет
То самый ближайший к нему новый, объединённый
Если не был
Если его ближайший сосед дальше, чем новый кластер
То самый ближайший к нему новый, объединённый
3.4) Добавляем новый кластер в список
На выходе получаем n кластеров
Второй шаг алгоритма.
Берётся весь датасет, и сканируется каждая его точка. Каждая точка присваивается к самому ближайшему кластеру (на основании расчета расстояния до всех его точек представителей)
Финальная кластеризация
1) Словарь для n кластеров
2) Для всех точек в массиве
Подбираем к какому классу принадлежит точка
Для всех кластеров
Минимальная дистанция = большое число
Минимальное расстояние от точки до кластера
Если дистанция меньше, чем минимальная
Минимальная дистанция = новая минимальная дистанция
Возвращаем индекс кластера, для которого дистанция минимальна
Присваиваем в словарь, в массив одного из классов
3) Возвращаем массивы индексов точек распределенные по классам
На выходе получаем кластеризованный набор данных.
Программная реализация на языке
Python с использованием библиотеки pyclustering.
import os
import random
import numpy as np
import matplotlib.pyplot as plt
def euclidean_distance(x, y):
return np.sqrt(np.sum((x - y) ** 2))
def get_representatives(data, k, fraction):
# Select k random points as initial representatives
representatives = data[np.random.choice(data.shape[0], size=k, replace=False), :]
# Find additional representatives by clustering with single linkage
while k < int(fraction * data.shape[0]):
dist_matrix = np.zeros((k, data.shape[0]))
for i in range(k):
for j in range(data.shape[0]):
dist_matrix[i][j] = euclidean_distance(representatives[i], data[j])
min_dist = np.min(dist_matrix, axis=0)
max_index = np.argmax(min_dist)
representatives = np.vstack((representatives, data[max_index]))
k += 1
return representatives
def get_clusters(data, representatives, alpha):
clusters = [[] for _ in range(representatives.shape[0])]
for i in range(data.shape[0]):
min_dist = np.inf
min_index = -1
for j in range(representatives.shape[0]):
dist = euclidean_distance(data[i], representatives[j])
if dist < min_dist:
min_dist = dist
min_index = j
clusters[min_index].append({'index': i, 'data': data[i]})
# Merge clusters that are closer than alpha
while True:
merged = False
for i in range(representatives.shape[0]):
for j in range(i + 1, representatives.shape[0]):
dist = euclidean_distance(representatives[i], representatives[j])
if dist < alpha:
merged = True
new_rep = (len(clusters) + len(clusters[j])) / (len(clusters[i]) + len(clusters[j])) * \
representatives[i] \
+ (len(clusters) + len(clusters[i])) / (len(clusters[i]) + len(clusters[j])) * \
representatives[j]
representatives[i] = new_rep
clusters[i] += clusters[j]
del clusters[j]
representatives = np.delete(representatives, j, axis=0)
break
if merged:
break
if not merged:
break
return clusters
if __name__ == '__main__':
# Generate random data
np.random.seed(0)
data = np.random.randn(100, 2)
# Clear previous console output
os.system('cls' if os.name == 'nt' else 'clear')
# Print initial Data
print(f'Data: {data}\n')
# Clustering parameters
k = 5
fraction = 0.1
alpha = 0.5
# Plot parameters
point_size = 15
# Cluster data using CURE
representatives = get_representatives(data, k, fraction)
clusters = get_clusters(data, representatives, alpha)
# Create a new figure and axis
fig, ax = plt.subplots()
# Print results
print(f'Representatives: {representatives}\n')
for i, cluster in enumerate(clusters):
print(f'Cluster {i}: {cluster}')
# Extract the points from the cluster dictionary
points = [p['data'] for p in cluster]
points = np.array(points)
# Generate a random color for the cluster
color = tuple(random.uniform(0, 1) for _ in range(3))
# Plot the points
ax.scatter(points[:, 0], points[:, 1], c=color, s=point_size, label=f'Cluster {i}')
# Set the axis labels and legend
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.legend()
# Show the plot
plt.show()
Результат работы программы:
Определим входные данные, которые мы будем использовать для нашего алгоритма кластеризации. Всего определяется 100 точек в двумерном пространстве.
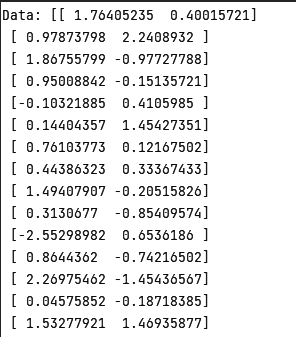
Далее из них выбирается несколько точек, которые будут поданы на вход алгоритма для начала кластеризации.
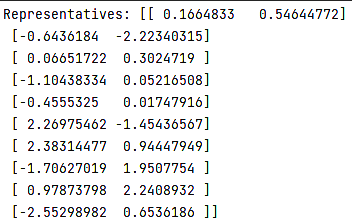
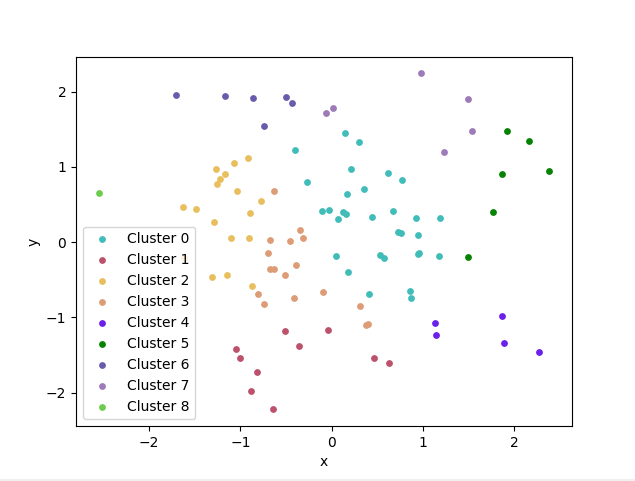
В результате видим список кластеров и точек, которые были к ним причислены

Визуальное отображение кластеров:
Источники:
-
https://digitrain.ru/articles/13812/ -
https://translated.turbopages.org/proxy_u/en-ru.ru.49047d43-63b9b6b3-9378cf2d-74722d776562/https/en.wikipedia.org/wiki/Cure_data_clustering -
https://algowiki-project.org/ru/Участник:JuliaA/Алгоритм_кластеризации_с_использованием_представлений -
https://russianblogs.com/article/6919119054/ -
https://www.ijirmf.com/wp-content/uploads/IJIRMF202006027.pdf